fviz_mca: Visualisation de l'Analyse des Correspondances Multiples - Logiciel R et analyse de données
Description
Graphique des variables individus/variables issues des r?sultats de l?Analyse des Correspondences Multiples (ACM).
Les fonctions suivantes, du package factoextra, sont utilis?e:
- fviz_mca_ind(): Graphique des individus
- fviz_mca_var(): Graphique des variables
- fviz_mca_biplot() (ou fviz_mca()): Biplot des individus et des variables
Installer et charger factoextra
Le package devtools est requis pour l?installation de factoextra.
if(!require(devtools)) install.packages("devtools")
devtools::install_github("kassambara/factoextra")Charger factoextra:
library("factoextra")Utilisation
# Graphique des individus
fviz_mca_ind(X, axes = c(1, 2),
geom = c("point", "text"), label = "all", invisible = "none",
labelsize = 4, pointsize = 2, habillage = "none",
addEllipses = FALSE, ellipse.level = 0.95, col.ind = "blue",
col.ind.sup = "darkblue", alpha.ind = 1, shape.ind = 19,
select.ind = list(name = NULL, cos2 = NULL, contrib = NULL),
map = "symmetric",
jitter = list(what = "label", width = NULL, height = NULL), ...)
# Graphique des variables
fviz_mca_var(X, axes = c(1, 2),
geom = c("point", "text"), label = "all",
invisible = "none", labelsize = 4, pointsize = 2, col.var = "red",
alpha.var = 1, shape.var = 17, col.quanti.sup = "blue",
col.quali.sup = "darkgreen", col.circle = "grey70",
select.var = list(name = NULL, cos2 = NULL, contrib = NULL),
map = "symmetric",
jitter = list(what = "label", width = NULL, height = NULL))
# Biplot des individus et des variables
fviz_mca_biplot(X, axes = c(1, 2), geom = c("point", "text"),
label = "all", invisible = "none", labelsize = 4, pointsize = 2,
habillage = "none", addEllipses = FALSE, ellipse.level = 0.95,
col.ind = "blue", col.ind.sup = "darkblue", alpha.ind = 1,
col.var = "red", alpha.var = 1, col.quanti.sup = "blue",
col.quali.sup = "darkgreen", shape.ind = 19, shape.var = 17,
select.var = list(name = NULL, cos2 = NULL, contrib = NULL),
select.ind = list(name = NULL, cos2 = NULL, contrib = NULL),
map = "symmetric", arrows = c(FALSE, FALSE),
jitter = list(what = "label", width = NULL, height = NULL), ...)
# Alias de fviz_mca_biplot()
fviz_mca(X, ...)Arguments
| Argument | Description |
|---|---|
| X | Un objet de class MCA [FactoMineR]; mca [ade4]. |
| axes | un vecteur num?rique de longueur 2 sp?cifiant les axes ? tracer. |
| geom | un texte sp?cifiant la g?om?trie ? utiliser pour le graphique. Les valeurs autoris?es sont la combinaison de c(?point?, ?arrow?, ?text?). Utilisez ?point? (pour afficher uniquement les points); ?text? pour afficher uniquement les ?tiquettes (texte d?annotation); c(?point?, ?text?) ou c(?arrow?, ?text?) pour montrer les deux types. |
| label | un vecteur de caract?res sp?cifiant les ?l?ments ? ?tiqueter. a valeur par d?faut est ?all?. Les valeurs autoris?es sont ?none? ou la combinaison de c(?ind?, ?ind.sup?,?var?, ?quali.sup?, ?quanti.sup?). Utilisez ?ind? pour ?tiqueter uniquement les individus actifs; ?ind.sup? est pour les individus suppl?mentaires. ?var? est pour les variables actives. ?quali.sup? est pour les variables qualitatives suppl?mentaires. ?quanti.sup? est pour les variables quantitatives suppl?mentaires. |
| invisible | une cha?ne de caract?re sp?cifiant les ?l?ments ? masquer sur le graphique. La valeur par d?faut est ?none?. Les valeurs autoris?es sont la combinaison de c(?ind?, ?ind.sup?,?var?, ?quali.sup?, ?quanti.sup?). |
| labelsize | taille de la police pour les ?tiquettes. |
| pointsize | la taille des points. |
| habillage | une variable cat?gorielle optionnelle de type ?factor. La valeur par d?faut est?none?. Si X est un objet de type MCA provenant de FactoMineR, habillage peut ?tre ?galement sp?cifi? par l?index ou le nom d?une variable qualitative suppl?mentaire. Cet argument est utilis? pour colorer les individus par des groupes (voir ?MCA dans FactoMineR). |
| addEllipses | valeur logique. Si TRUE, ajoute des ellipses autour des individus lorsque habillage!= ?none?. |
| ellipse.level | la taille de l?ellipse de concentration en probabilit? normale (i.e.: 0.95, 0.68, ?). |
| col.ind,col.var | couleurs pour les individus et les variables, respectivement. Les valeurs possibles comprennent ?galement: ?cos2?, ?contrib?, ?coord?, ?x? or ?y?. Dans ce cas, les couleurs des individus/variables sont automatiquement contr?l?es par leurs qualit?s (?cos2?), leurs contributions (?contrib?), leurs coordonn?es (x^2 + y^2, ?coord?), les valeurs x (?x?) ou les valeurs y (?y?). Pour utiliser la coloration automatique (par cos2, contrib, ?.), assurez-vous que habillage = ?none?. |
| col.ind.sup | couleur des individus suppl?mentaires. |
| alpha.ind,alpha.var | contr?le la transparence des couleurs des individus et des variables, respectivement. La valeur peut varier de 0 (transparence totale) ? 1 (aucune transparence). La valeur par d?faut est 1. Les valeurs admises comprennent ?galement: ?cos2?, ?contrib?, ?coord?, ?x? ou ?y?, comme pour les arguments col.ind et col.var. Pour utiliser ceux-ci, assurez-vous que habillage = ?none?. |
| select.ind,select.var |
S?lection des individus/variables ? dessiner. Les valeurs autoris?es sont NULL ou une liste contenant le nom des arguments, cos2 ou contrib. La valeur par d?faut est list(name = NULL, cos2 = NULL, contrib = NULL):
|
| map | cha?ne de caract?res sp?cifiant le type de graphique. Les options autoris?es incluent: ?rowprincipal? ?symetric?, ?colprincipal?, ?symbiplot?, ?rowgab?, ?colgab?, ?rowgreen? et ?colgreen?. Voir la section d?tails |
| jitter | Un param?tre d?ajustement de la position des points et textes pour r?duire le chevauchement. C?est une liste contenant les objets what, width et height (Ex.; jitter = list(what, width, height)). what: l??l?ment ? ajuster. Les valeurs possibles sont ?point? ou ?p?; ?label? ou ?l?; ?both? ou ?b?. width: degr? d?ajustement selon l?axe des x (ex: 0.2). height: degr? d ?ajustement selon l?axe des y (ex: 0.2). |
| col.quanti.sup, col.quali.sup | couleur pour les variables quantitative/qualitative suppl?mentaires. |
| arrows | Vecteur logique de longueur deux, pr?cisant si le graphique doit contenir des points (FALSE, par d?faut) ou fl?ches (TRUE). La premi?re valeur s?applique aux variables lignes et la deuxi?me valeur s?applique aux variables colonnes. |
| ? | Arguments optionnels ? passer ? la fonction fviz_mca_biplot(). |
Details
Le graphique par d?faut de l?ACM (analyse des correspondances multiples) est un graphique ?sym?trique?, dans lequel les lignes et les colonnes sont repr?sent?es en coordonn?es principales. Dans cette situation, il est impossible d?interpr?ter la distance entre les points lignes et les points colonnes. Pour surmonter ce probl?me, le plus simple est de faire un graphique asym?trique. Cela signifie que, les profils colonnes doivent ?tre repr?sent?es dans l?espace des lignes ou vice-versa. Les options autoris?es pour l?argument map sont:
?rowprincipal? or ?colprincipal?: graphique asym?triques avec soit des lignes en coordonn?es principales et les colonnes en coordonn?es standards, ou vice versa. Ces graphiques conservent respectivement la m?trique des lignes ou celle des colonnes.
?symbiplot?: Les lignes et les colonnes sont transform?es afin d?avoir des variances ?gales aux valeurs singuli?res (racines carr?s des valeurs propres), ce qui donne un biplot sym?trique mais ne pr?serve pas les m?triques des lignes ou des colonnes.
?rowgab? ou ?colgab?: graphiques asym?triques, propos?es par Gabriel & Odoroff (1990), avec des lignes (respectivement colonnes) en coordonn?es principales et colonnes (respectivement lignes) en coordonn?es standards multipli? par la masse du point correspondant.
?rowgreen? ou ?colgreen?: graphiques appel?s aussi ?biplots de contribution?, montrant visuellement les points les plus contributifs (Greenacre 2006b). Ils sont semblables ? ?rowgab? et ?colgab?, sauf que les points en coordonn?es standard sont multipli?s par la racine carr?e des masses correspondantes, donnant les reconstructions des r?sidus standardis?s.
Valeur
Un graphique de type ggplot2
Exemples
Analyse des correspondences multiples
Une Analyse des Correspondences Multiples (ACM) est effectu?e en utilisant la fonction MCA() [dans FactoMineR] et les donn?es poison [dans FactoMineR]:
# Installer et charger FactoMineR pour effectuer une MCA
# install.packages("FactoMineR")
library("FactoMineR")
data(poison)
poison.active <- poison[1:55, 5:15]
head(poison.active[, 1:6]) Nausea Vomiting Abdominals Fever Diarrhae Potato
1 Nausea_y Vomit_n Abdo_y Fever_y Diarrhea_y Potato_y
2 Nausea_n Vomit_n Abdo_n Fever_n Diarrhea_n Potato_y
3 Nausea_n Vomit_y Abdo_y Fever_y Diarrhea_y Potato_y
4 Nausea_n Vomit_n Abdo_n Fever_n Diarrhea_n Potato_y
5 Nausea_n Vomit_y Abdo_y Fever_y Diarrhea_y Potato_y
6 Nausea_n Vomit_n Abdo_y Fever_y Diarrhea_y Potato_yres.mca <- MCA(poison.active, graph=FALSE)fviz_mca_ind(): Graphique des individus
# Graphique par d?faut
fviz_mca_ind(res.mca)
# Changer le titre principal et celui des axes
fviz_mca_ind(res.mca) +
labs(title = "MCA", x = "Dim.1", y ="Dim.2" )
# Changer les limites des axes en sp?cifiant le min et le max
fviz_mca_ind(res.mca) +
xlim(-0.8, 1.5) + ylim (-1.5, 1.5)
# Utiliser seulement du texte
fviz_mca_ind(res.mca, geom = "text")
# Utiliser uniquement des points
fviz_mca_ind(res.mca, geom="point")
# Changer la taille des points
fviz_mca_ind(res.mca, geom="point", pointsize = 4)
# Changer la couleur des points et le th?me
fviz_mca_ind(res.mca, col.ind = "blue")+
theme_minimal()
# R?duire le ch?vauchement
fviz_mca_ind(res.mca,
jitter = list(width = 0.2, height = 0.2))
# Contr?ler automatiquement la couleurs des individus
# par les valeurs de cos2 ou de contributions
# cos2 = qualit? de r?pr?sentation sur le graphique
fviz_mca_ind(res.mca, col.ind="cos2")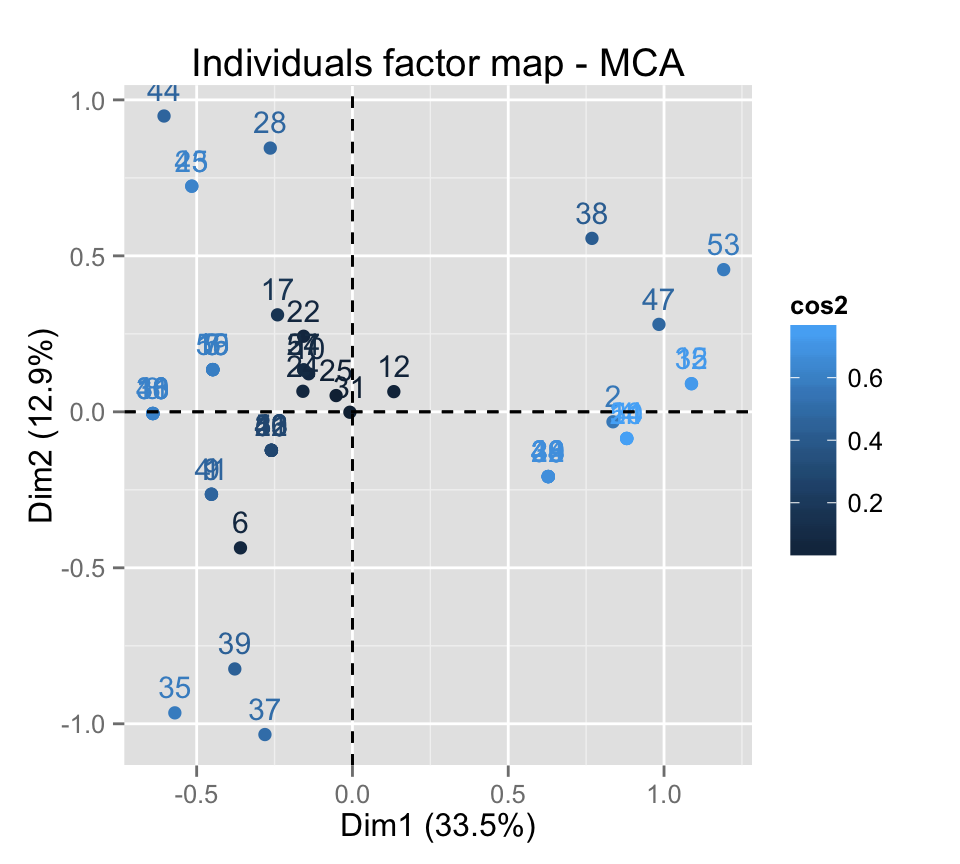
# Gradient de couleur
fviz_mca_ind(res.mca, col.ind="cos2") +
scale_color_gradient2(low="white", mid="blue",
high="red", midpoint=0.4, space = "Lab")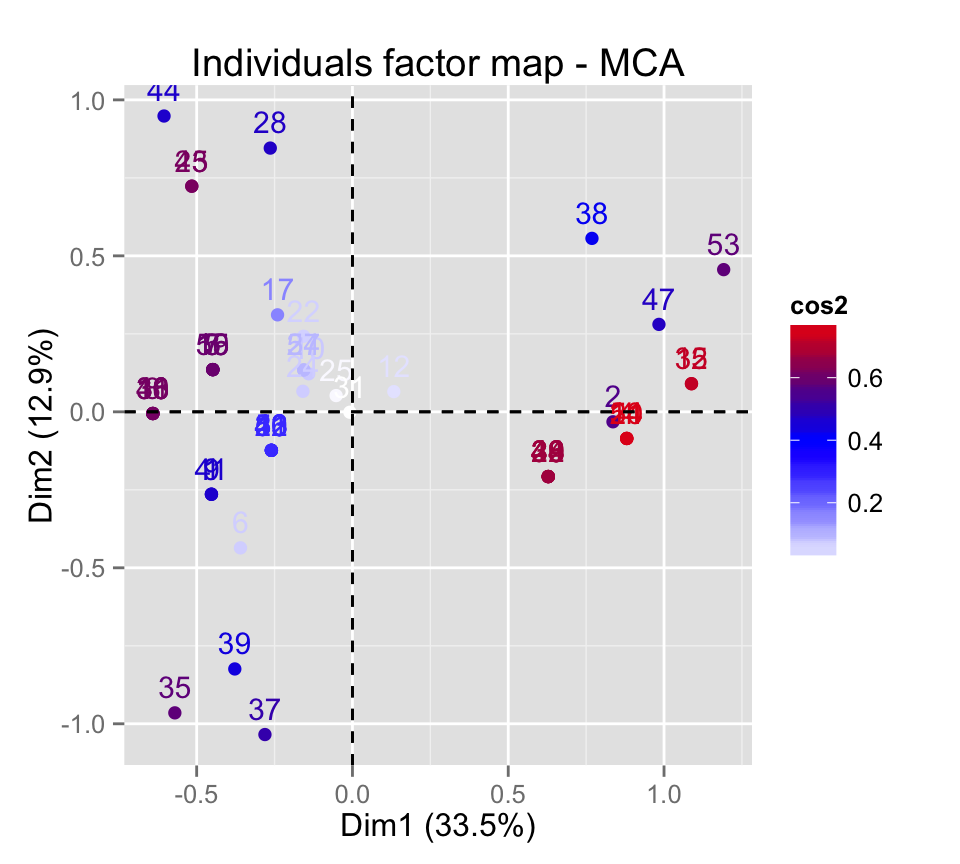
# Changer le th?me et utiliser uniquement des points
fviz_mca_ind(res.mca, col.ind="cos2", geom = "point") +
scale_color_gradient2(low="white", mid="blue",
high="red", midpoint=0.4, space = "Lab")+ theme_minimal()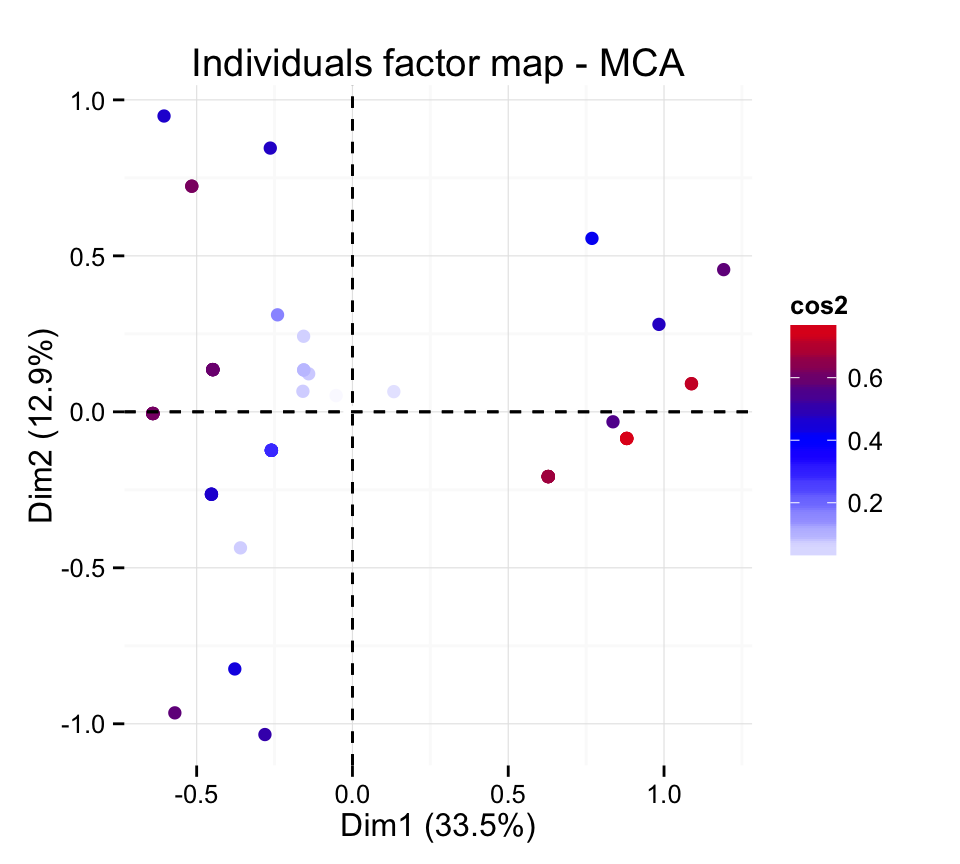
# Colorer en fonction de la contribution
fviz_mca_ind(res.mca, col.ind="contrib") +
scale_color_gradient2(low="white", mid="blue",
high="red", midpoint=1.5, space = "Lab")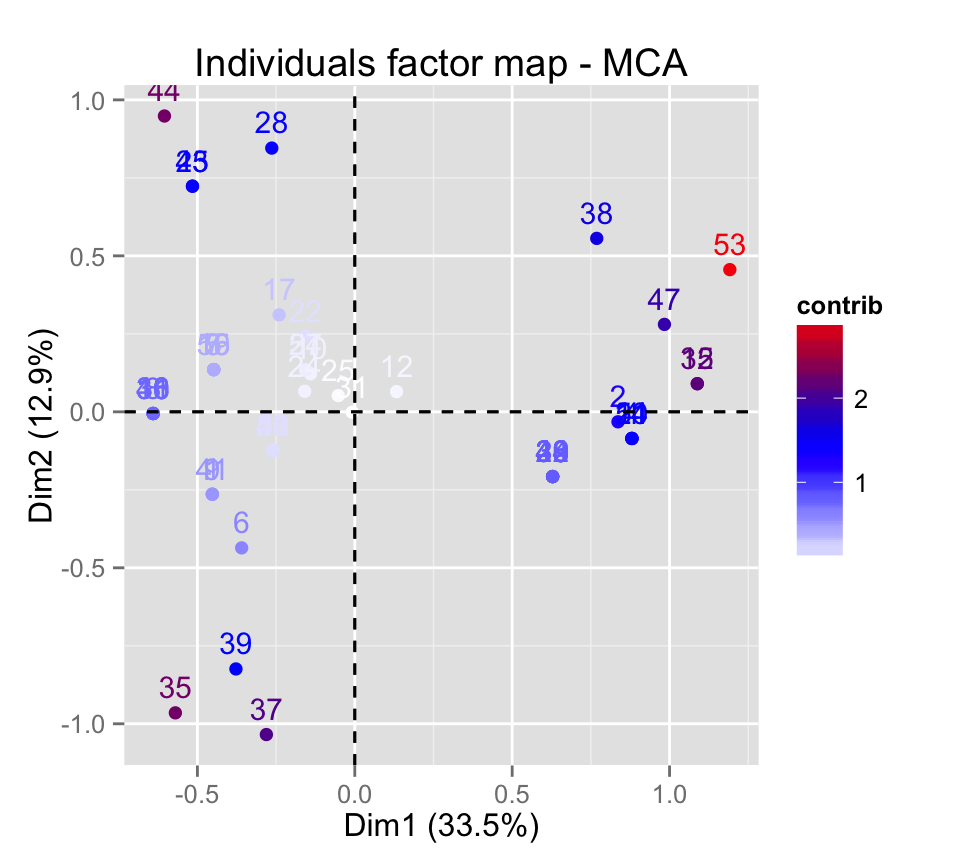
# Contr?ler la transparence des couleurs
# en fonction de la contribution
fviz_mca_ind(res.mca, alpha.ind="contrib") +
theme_minimal()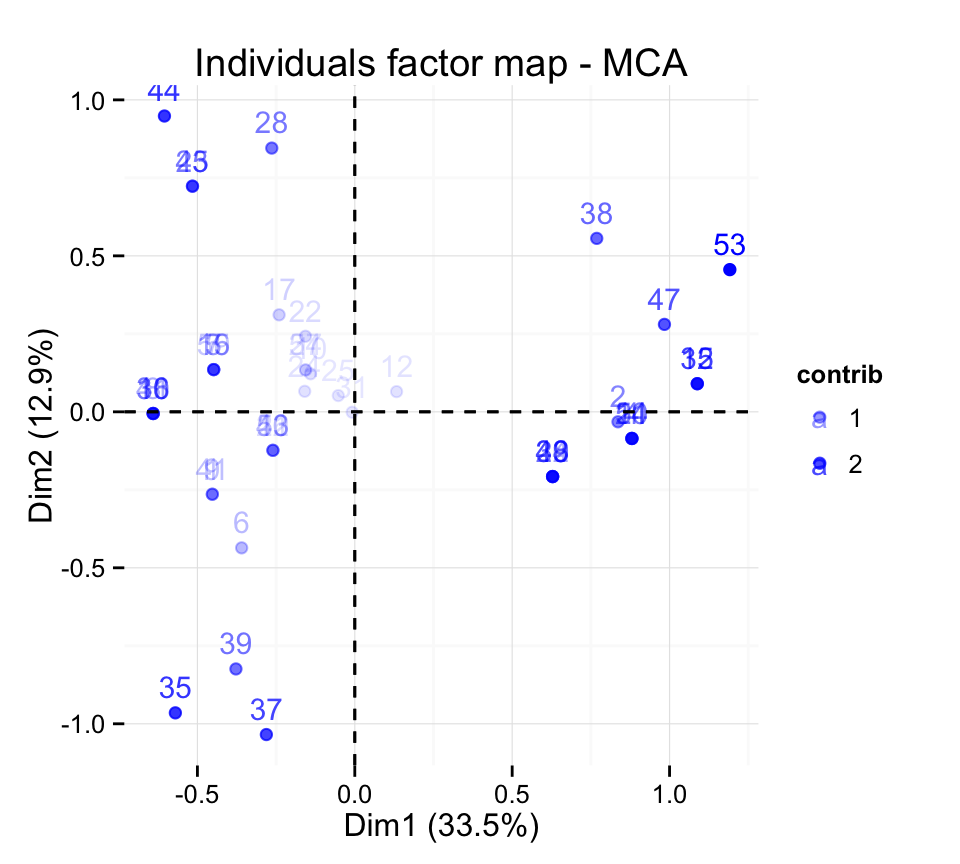
# Colorer les individus par groupes
grp <- as.factor(poison.active[, "Vomiting"])
fviz_mca_ind(res.mca, label="none", habillage=grp)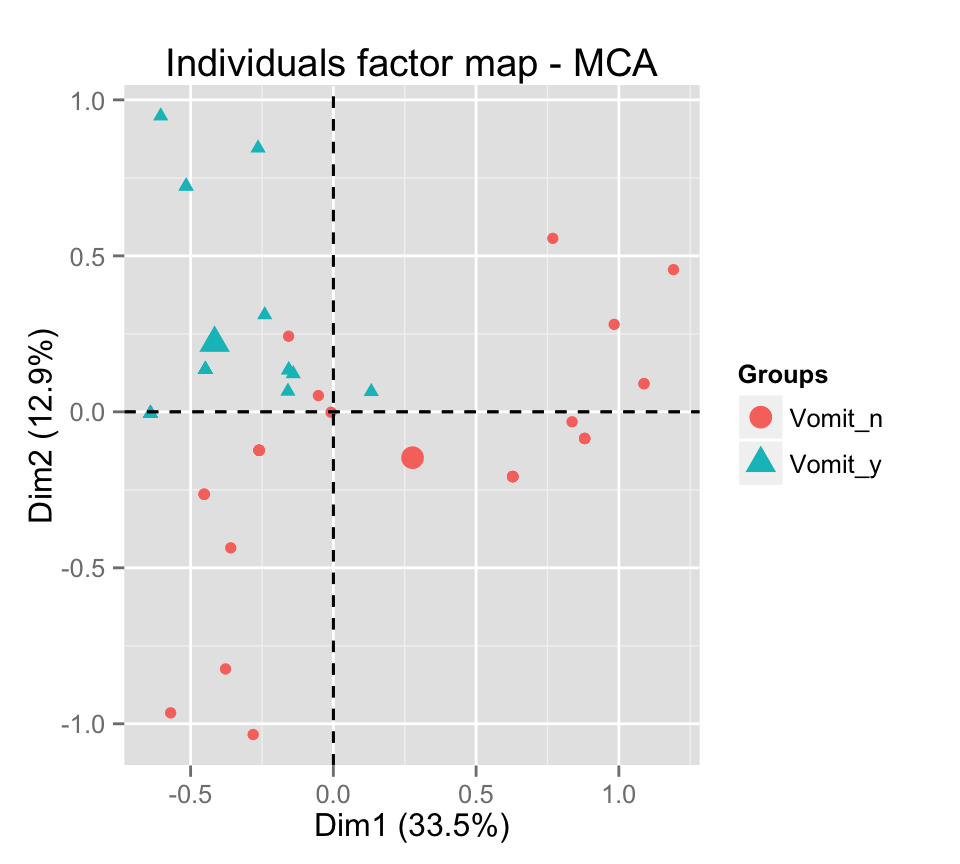
# Ajouter des ellipses
p <- fviz_mca_ind(res.mca, label="none", habillage=grp,
addEllipses=TRUE, ellipse.level=0.95)
print(p)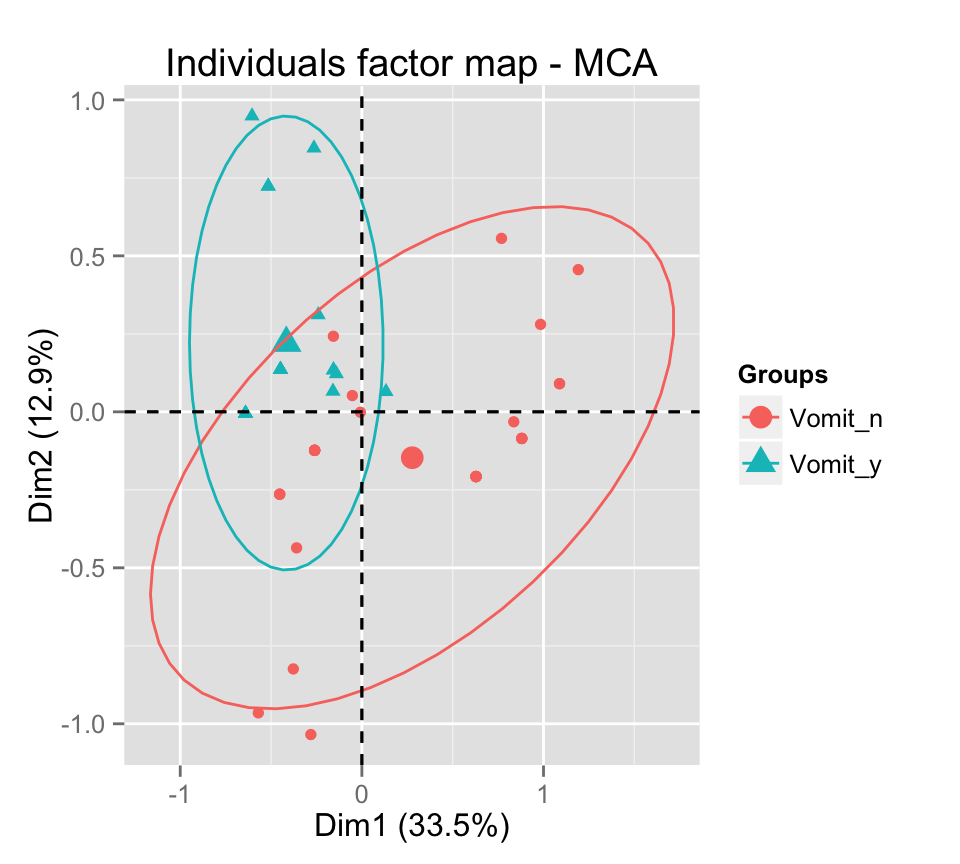
# Changer la couleur des groupes en utilisant
# les palettes RColorBrewer
p + scale_color_brewer(palette="Dark2") +
theme_minimal()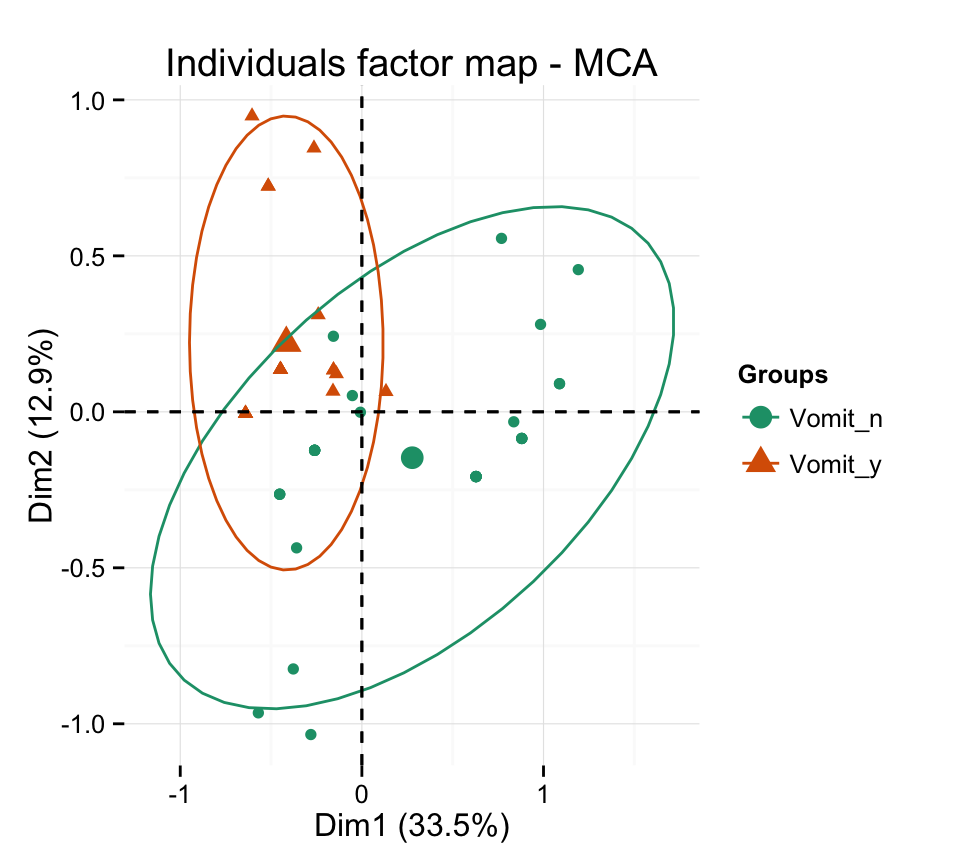
p + scale_color_brewer(palette="Paired") +
theme_minimal()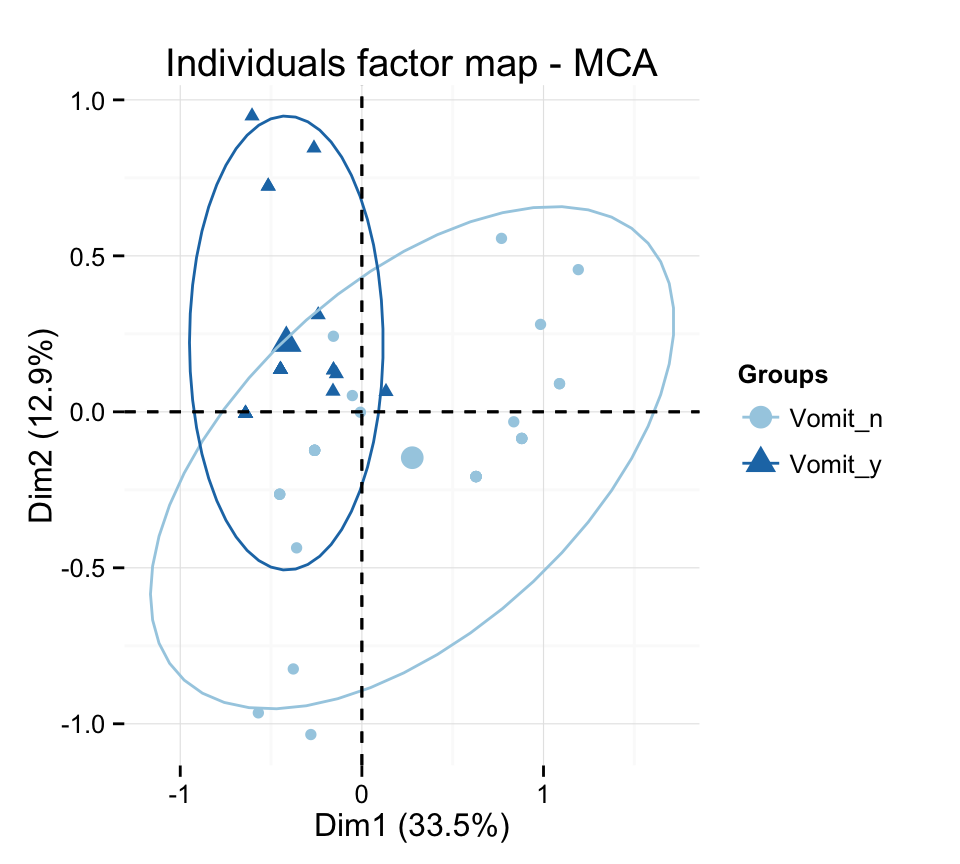
p + scale_color_brewer(palette="Set1") +
theme_minimal()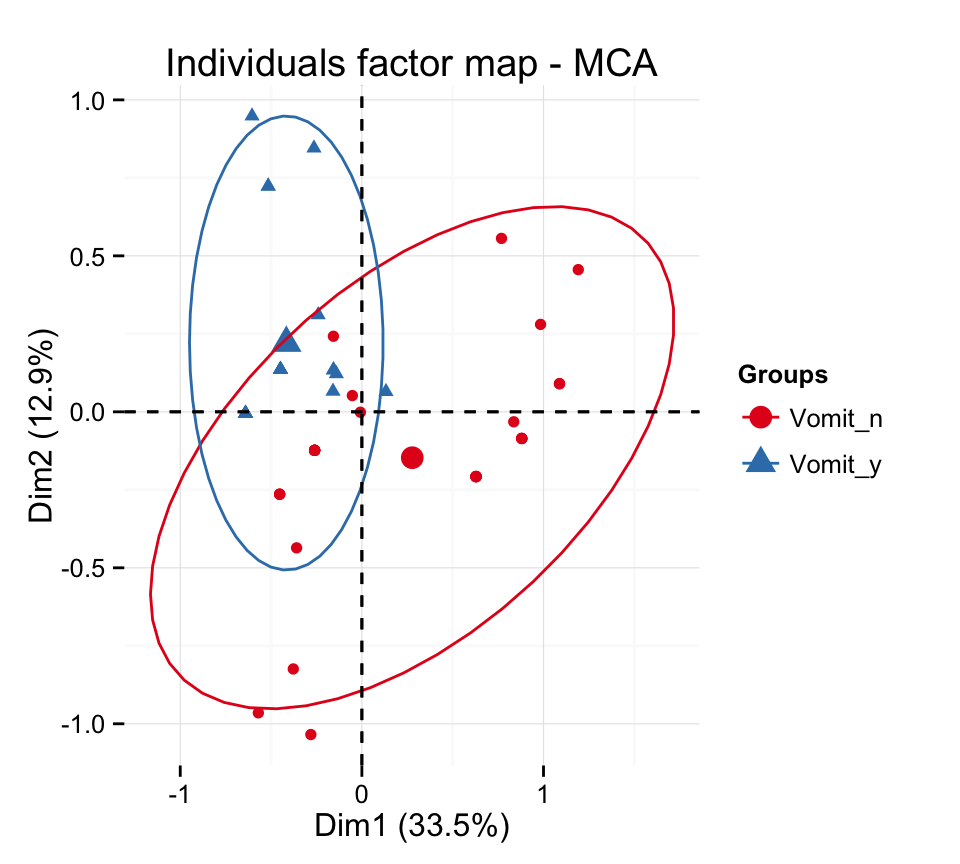
# Changer la couleur manuellement
p + scale_color_manual(values=c("#999999", "#E69F00", "#56B4E9"))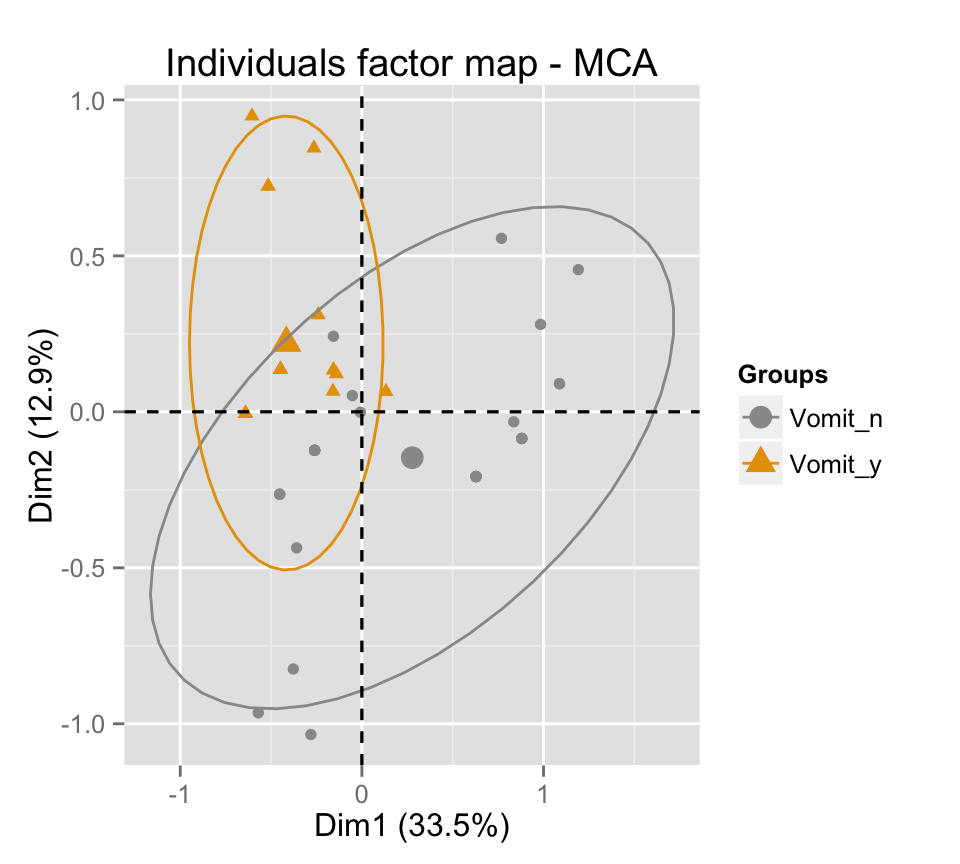
# S?lectionner et visualiser les individus avec cos2 >= 0.4
fviz_mca_ind(res.mca, select.ind = list(cos2 = 0.4))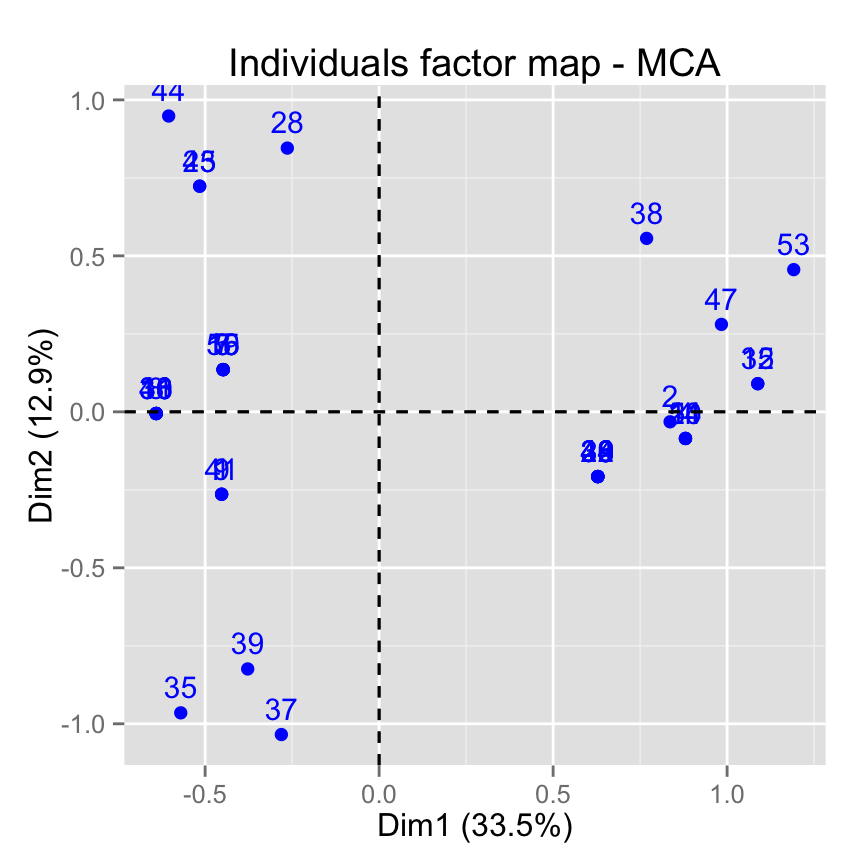
# S?lectionner le top 20 selon le cos2
fviz_mca_ind(res.mca, select.ind = list(cos2 = 20))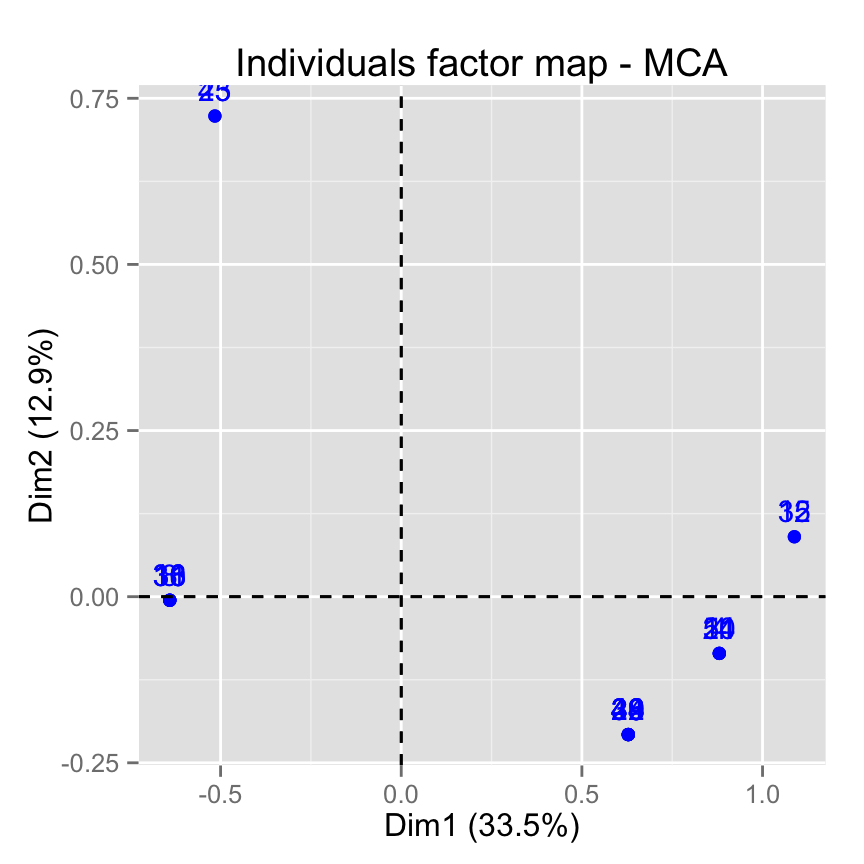
# Selectionner le top 20 selon la contribution
fviz_mca_ind(res.mca, select.ind = list(contrib = 20))
# S?lectionner par le nom
fviz_mca_ind(res.mca,
select.ind = list(name = c("44", "38", "53", "39")))
fviz_mca_var(): Graphique des variables
# Graphique par d?faut
fviz_mca_var(res.mca)
# Changer la couleur et le th?me
fviz_mca_var(res.mca, col.var="steelblue")+
theme_minimal()
# Contr?ler la couleur selon la contribution
fviz_mca_var(res.mca, col.var = "contrib")+
scale_color_gradient2(low = "white", mid = "blue",
high = "red", midpoint = 2) +
theme_minimal()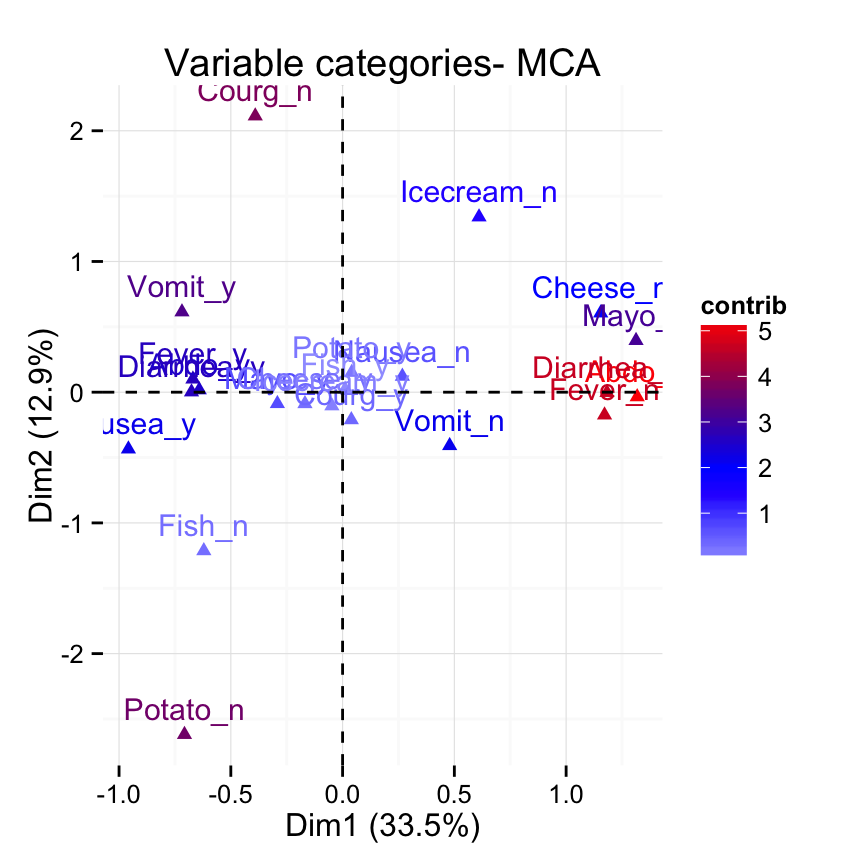
# Contr?ler la transparence des variables
# selon leurs contributions
fviz_mca_var(res.mca, alpha.var = "contrib") +
theme_minimal()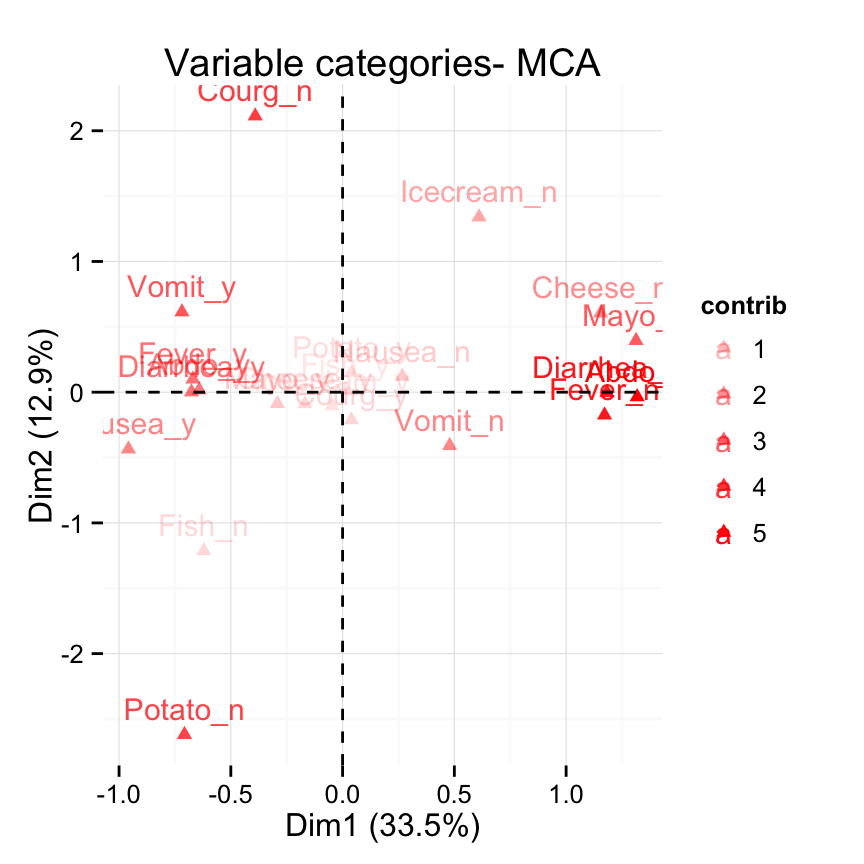
# S?lectionner et visualiser les variables avec cos2 >= 0.4
fviz_mca_var(res.mca, select.var = list(cos2 = 0.4))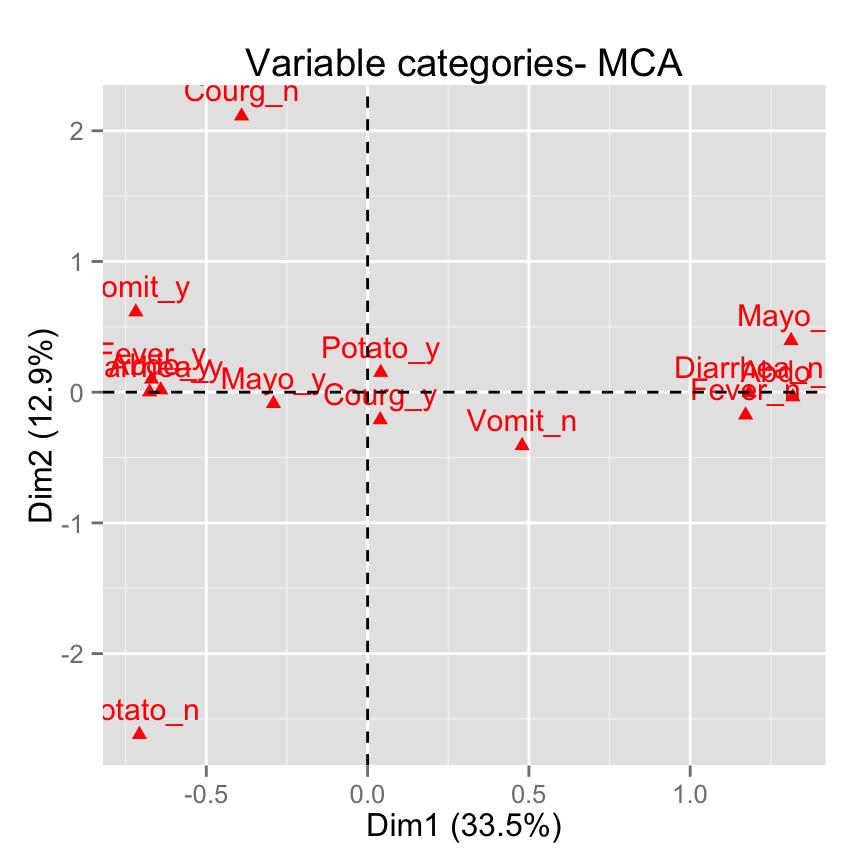
# S?lectionner le top 10 selon la contribution
fviz_mca_var(res.mca, select.var = list(contrib = 10))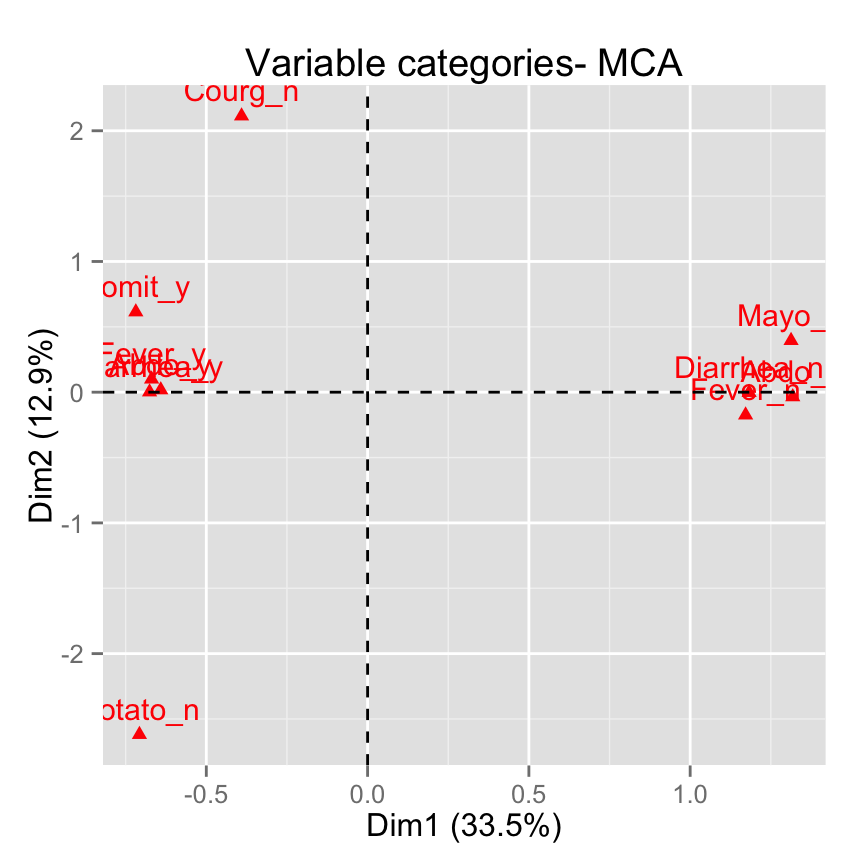
# S?lectionner par noms
fviz_mca_var(res.mca,
select.var= list(name = c("Courg_n", "Fever_y", "Fever_n")))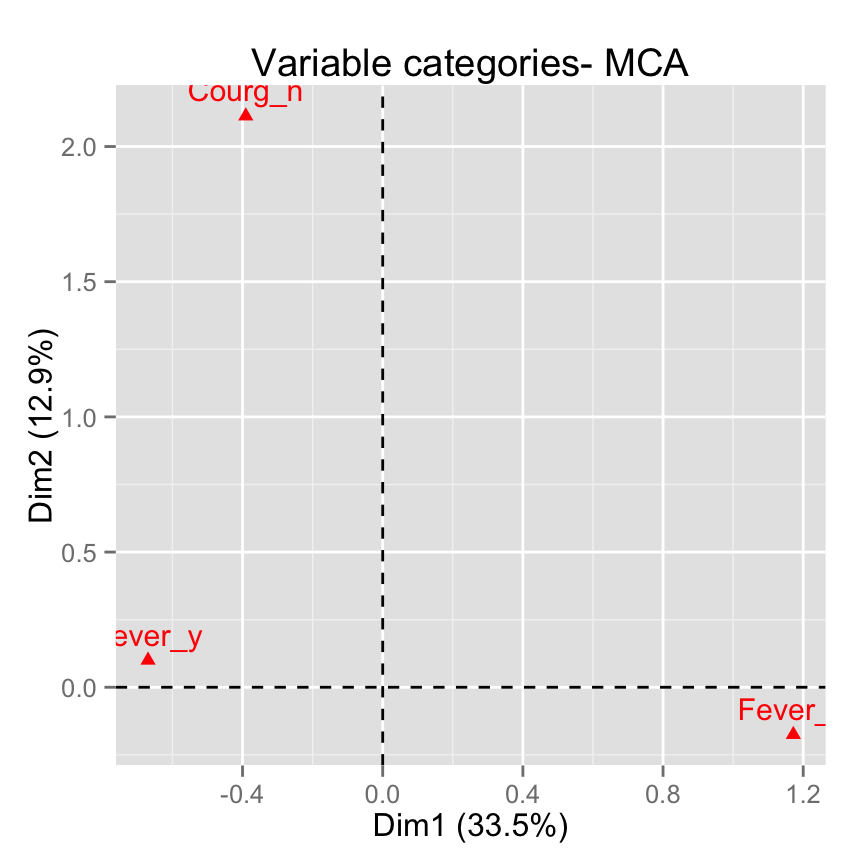
fviz_mca_biplot(): Biplot des individus et des variables
fviz_mca_biplot(res.mca)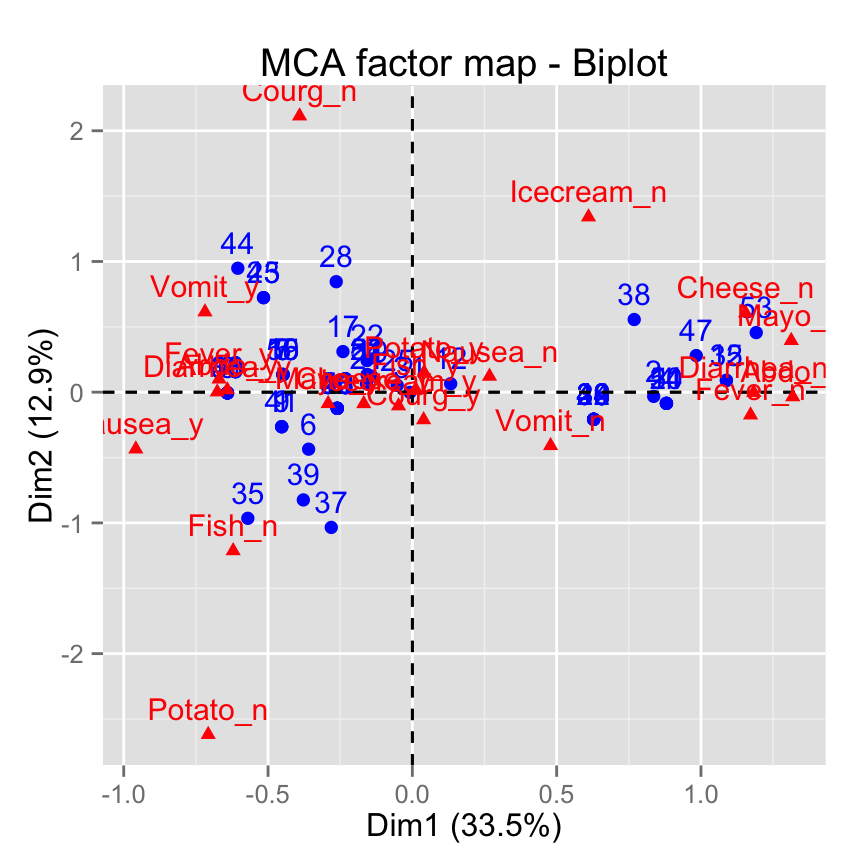
# Annoter uniquement les variables
fviz_mca_biplot(res.mca, label ="var")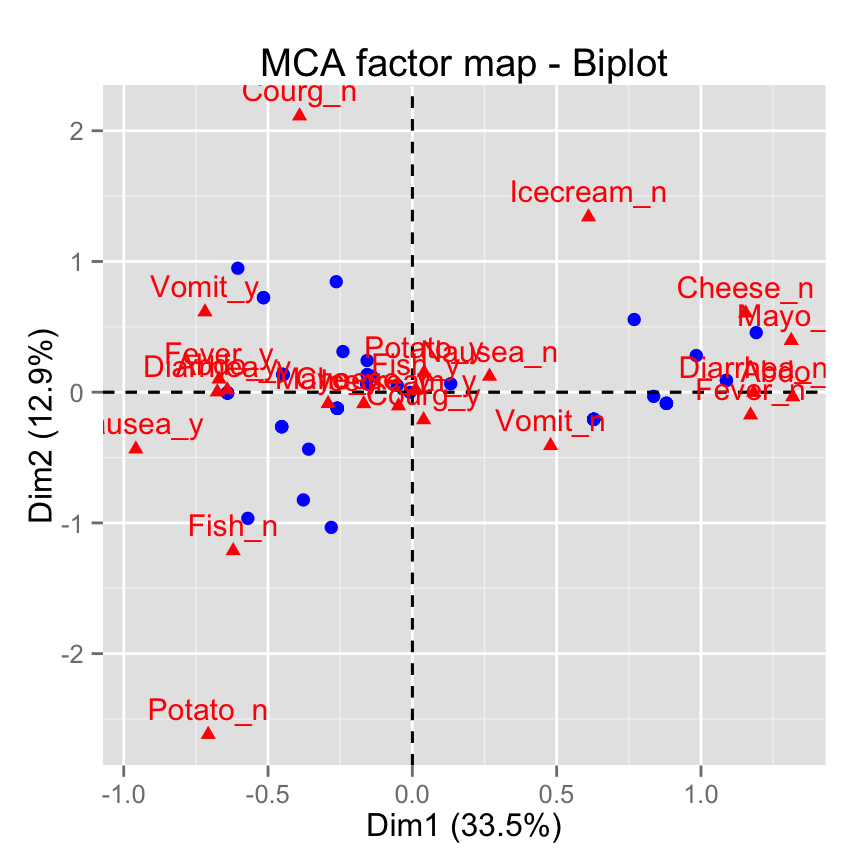
# Annoter uniquement les individus
fviz_mca_biplot(res.mca, label ="ind")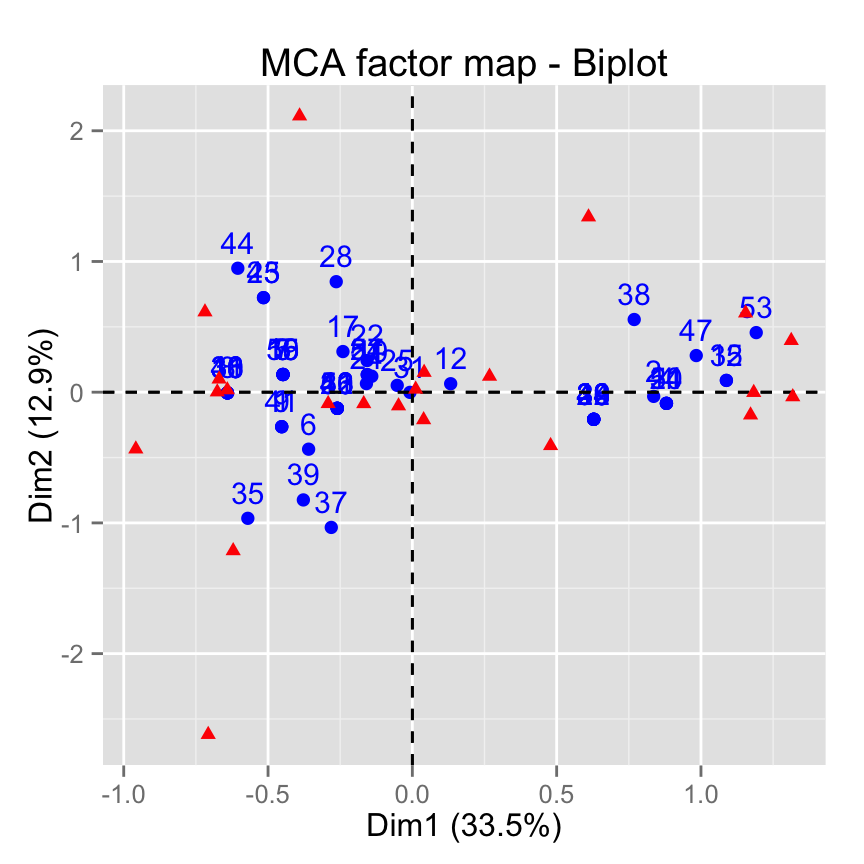
# Cacher les variables
fviz_mca_biplot(res.mca, invisible ="var")
# Cacher les individus
fviz_mca_biplot(res.mca, invisible ="ind")
# Contr?ler la couleur des individus selon le cos2
fviz_mca_biplot(res.mca, label ="var", col.ind="cos2") +
theme_minimal()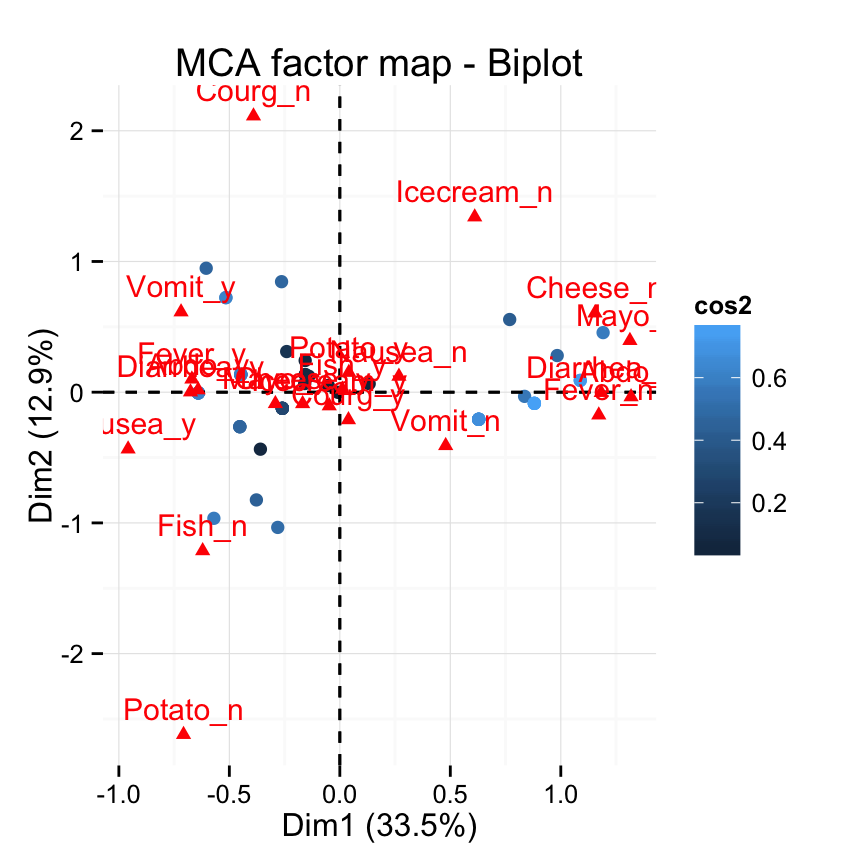
# Change la couleur par groups, ajouter des ellipses
fviz_mca_biplot(res.mca, label="var", col.var ="blue",
habillage=grp, addEllipses=TRUE, ellipse.level=0.95) +
theme_minimal()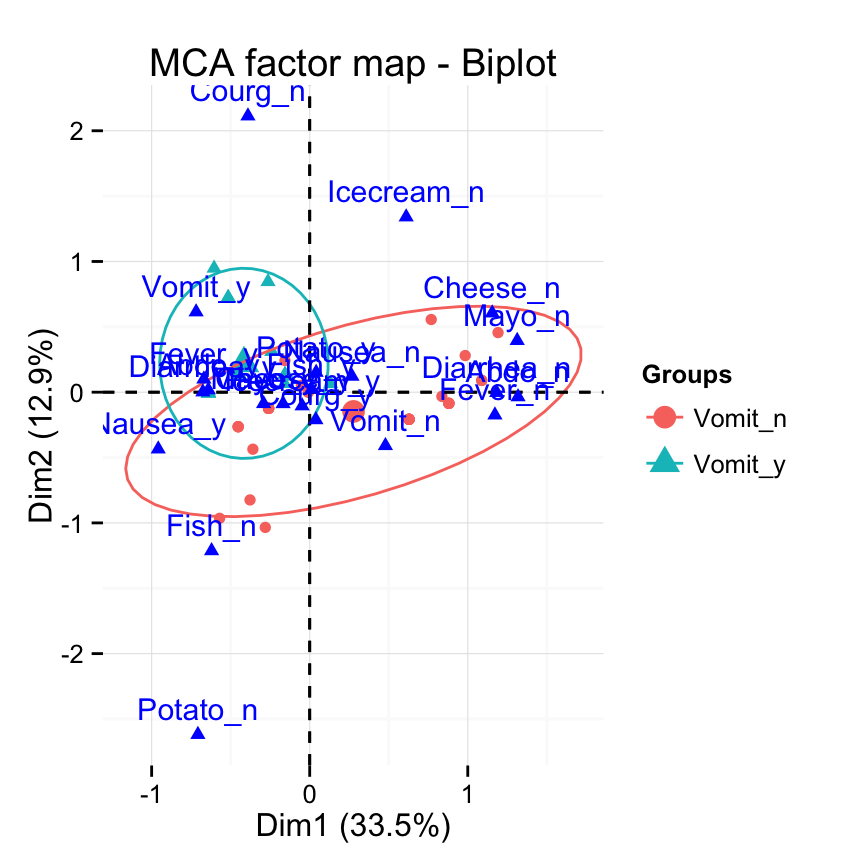
# Top 30 des individus les plus contributifs
# et top 10 des variables les plus contributifs
fviz_mca_biplot(res.mca,
select.ind = list(contrib = 30),
select.var = list(contrib = 10))
Infos
Cette analyse a ?t? r?alis?e en utilisant le logiciel R (ver. 3.2.1) et factoextra (ver. 1.0.3)
Show me some love with the like buttons below... Thank you and please don't forget to share and comment below!!
Montrez-moi un peu d'amour avec les like ci-dessous ... Merci et n'oubliez pas, s'il vous plaît, de partager et de commenter ci-dessous!
Recommended for You!
Click to follow us on Facebook:
Comment this article by clicking on "Discussion" button (top-right position of this page)







