Matrice de corrélation: Formattage et visualisation
Le calcul d?une matrice de corr?lation est d?crit en d?tails ici. L?objectif de cet article est double. D?une part, nous allons utiliser le package Hmisc de R pour produire une table des coefficients de corr?lation ainsi que les p-values correspondantes. D?autre part, nous allons voir comment formatter cette matrice de corr?lation en un tableau de 4 colonnes contenant le nom des lignes de la matrice et celui des colonnes, les coefficients de corr?lation et les p-values des corr?lations.
Donn?es
La table de donn?es mtcars disponible dans R est utilis?e dans les exemples suivants :
mydata <- mtcars[, c(1,3,4,5,6,7)]
head(mydata) mpg disp hp drat wt qsec
Mazda RX4 21.0 160 110 3.90 2.620 16.46
Mazda RX4 Wag 21.0 160 110 3.90 2.875 17.02
Datsun 710 22.8 108 93 3.85 2.320 18.61
Hornet 4 Drive 21.4 258 110 3.08 3.215 19.44
Hornet Sportabout 18.7 360 175 3.15 3.440 17.02
Valiant 18.1 225 105 2.76 3.460 20.22Calcul de la matrice de corr?lation
La fonction rcorr() du package Hmisc peut ?tre utilis?e. Elle offre la possibilit? de faire les tests de corr?lation de Pearson et de Spearman.
library(Hmisc)
res<-rcorr(as.matrix(mydata))Comme r?sultat, la fonction rcorr() renvoie une liste incluant les ?l?ments suivants : - r : la matrice de corr?lation. - P : les p-values correspondant aux niveaux de significativit? des corr?lations.
# Affichage de la matrice de corr?lation
signif(res$r, 2) mpg disp hp drat wt qsec
mpg 1.00 -0.85 -0.78 0.680 -0.87 0.420
disp -0.85 1.00 0.79 -0.710 0.89 -0.430
hp -0.78 0.79 1.00 -0.450 0.66 -0.710
drat 0.68 -0.71 -0.45 1.000 -0.71 0.091
wt -0.87 0.89 0.66 -0.710 1.00 -0.170
qsec 0.42 -0.43 -0.71 0.091 -0.17 1.000# Affichage des p-values des corr?lations
signif(res$P,2) mpg disp hp drat wt qsec
mpg NA 9.4e-10 1.8e-07 1.8e-05 1.3e-10 1.7e-02
disp 9.4e-10 NA 7.1e-08 5.3e-06 1.2e-11 1.3e-02
hp 1.8e-07 7.1e-08 NA 1.0e-02 4.1e-05 5.8e-06
drat 1.8e-05 5.3e-06 1.0e-02 NA 4.8e-06 6.2e-01
wt 1.3e-10 1.2e-11 4.1e-05 4.8e-06 NA 3.4e-01
qsec 1.7e-02 1.3e-02 5.8e-06 6.2e-01 3.4e-01 NAFormattage de la matrice de corr?lation en 4 colonnes
La fonction personnalis?e suivante est utilis?e :
# ++++++++++++++++++++++++++++
# flattenCorrMatrix
# ++++++++++++++++++++++++++++
# cormat : matrice des coefficients de corr?lation
# pmat : matrice des p-values
flattenCorrMatrix <- function(cormat, pmat) {
ut <- upper.tri(cormat)
data.frame(
row = rownames(cormat)[row(cormat)[ut]],
column = rownames(cormat)[col(cormat)[ut]],
cor =(cormat)[ut],
p = pmat[ut]
)
}La fonction flattenCorrMatrix va formatter la matrice de corr?lation en un tableau de 4 colonnes : row, column, coefficient de corr?lation et p-value.
Le tableau final contient les p-values et les coefficients de corr?lation entre chaque variable et les autres.
flattenCorrMatrix(res$r, res$P) row column cor p
1 mpg disp -0.8476 9.380e-10
2 mpg hp -0.7762 1.788e-07
3 disp hp 0.7909 7.143e-08
4 mpg drat 0.6812 1.776e-05
5 disp drat -0.7102 5.282e-06
6 hp drat -0.4488 9.989e-03
7 mpg wt -0.8677 1.294e-10
8 disp wt 0.8880 1.222e-11
9 hp wt 0.6587 4.146e-05
10 drat wt -0.7124 4.784e-06
11 mpg qsec 0.4187 1.708e-02
12 disp qsec -0.4337 1.314e-02
13 hp qsec -0.7082 5.766e-06
14 drat qsec 0.0912 6.196e-01
15 wt qsec -0.1747 3.389e-01Visualisation de la matrice de correlation
Vous pouvez utiliser la fonction chart.Correlation() du package PerformanceAnalytics pour une visualisation graphique de la matrice de corr?lation.
Les histogrammes de distribution des variables sont montr?s sur la diagonale. Les ast?riques indiquent le niveau de significativit? de la corr?lation. Pour chaque seuil de significativit? on a un symbole correspondant :
p-values(0, 0.001, 0.01, 0.05, 0.1, 1) <=> symboles(?***?, ?**?, ?*?, ?.?, " ?)
library(PerformanceAnalytics)
chart.Correlation(mydata, histogram=TRUE, pch=19)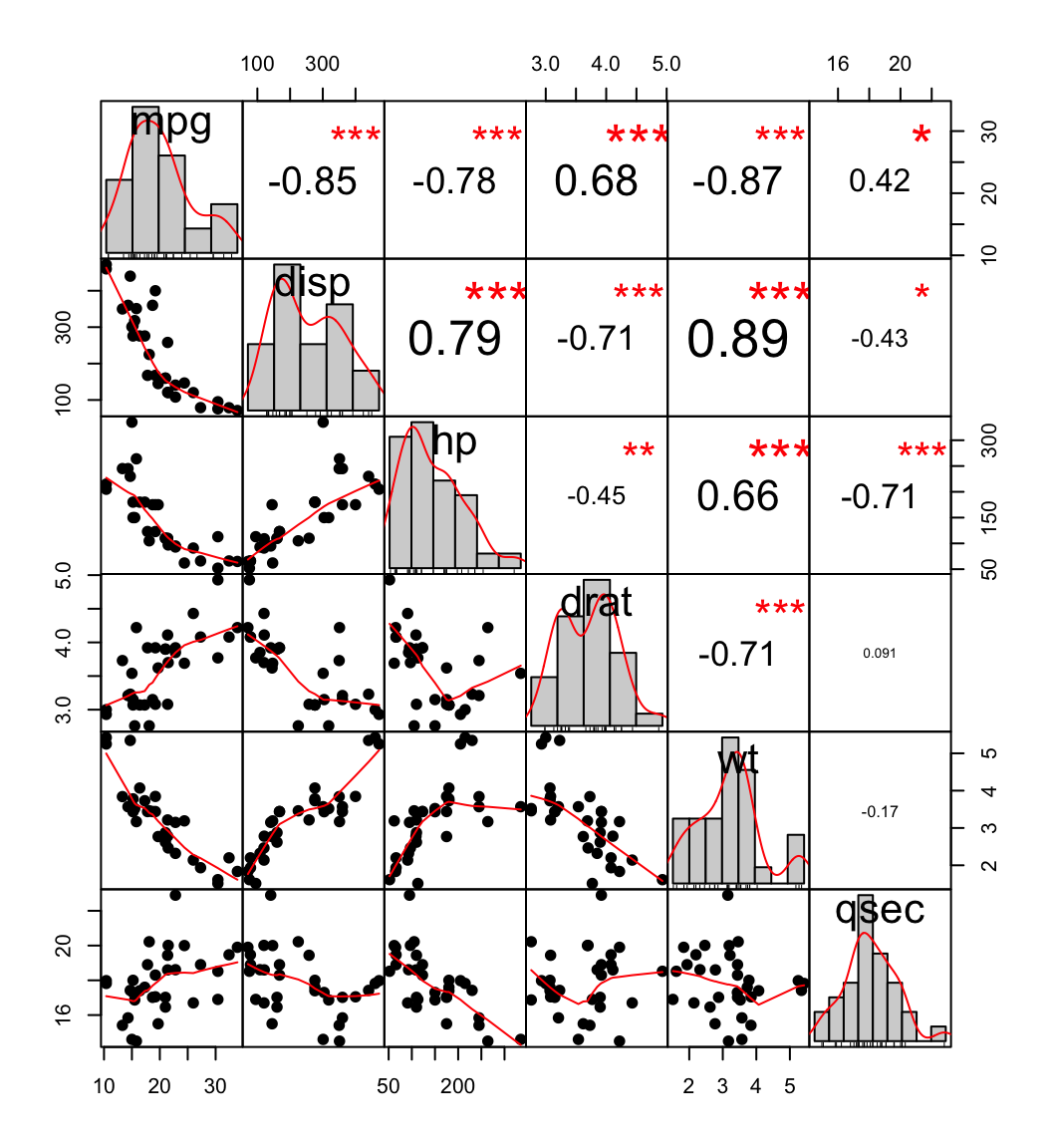
Infos
Cette analyse a ?t? faite avec R (ver. 3.1.0).
Show me some love with the like buttons below... Thank you and please don't forget to share and comment below!!
Montrez-moi un peu d'amour avec les like ci-dessous ... Merci et n'oubliez pas, s'il vous plaît, de partager et de commenter ci-dessous!
Recommended for You!
Click to follow us on Facebook:
Comment this article by clicking on "Discussion" button (top-right position of this page)







