ggplot2 échelle et transformation des axes - Logiciel R et visualisation de données
Ce tutoriel R d?crit comment modifier les limites des axes x et y (valeurs minimales et maximales) en utilisant le package ggplot2. La transformation des axes (?chelle logarithmique, racine carr?e, ?) et les axes pour des dates sont ?galement couverts dans cet article.
Pr?parer les donn?es
Le jeu de donn?es ToothGrowth est utilis? dans les exemples ci-dessous :
# Convertir la variable dose de type "numeric" au type "factor"
ToothGrowth$dose <- as.factor(ToothGrowth$dose)
head(ToothGrowth)## len supp dose
## 1 4.2 VC 0.5
## 2 11.5 VC 0.5
## 3 7.3 VC 0.5
## 4 5.8 VC 0.5
## 5 6.4 VC 0.5
## 6 10.0 VC 0.5Assurez-vous que la variable dose soit convertie en une variable de type ?factor? en utilisant le script de R ci-dessus.
Exemple de graphes
library(ggplot2)
# Box plot
bp <- ggplot(ToothGrowth, aes(x=dose, y=len)) + geom_boxplot()
bp
# Nuage de points
sp <- ggplot(cars, aes(x = speed, y = dist)) + geom_point()
sp
Changer les limites des axes x et y
Il existe diff?rentes fonctions pour changer les limites des axes:
- xlim() et ylim()
- expand_limits()
- scale_x_continuous() et scale_y_continuous()
Utiliser les fonctions xlim() et ylim()
Pour changer les limites d?un axe continu, les fonctions xlim() et ylim() peuvent ?tre utilis?es comme suit :
# Limites de l'axe des x
sp + xlim(min, max)
# Limites de l'axe des y
sp + ylim(min, max)min et max sont les valeurs minimales et maximales de chaque axe.
# Box plot : changer les limites de l'axe des y
bp + ylim(0,50)
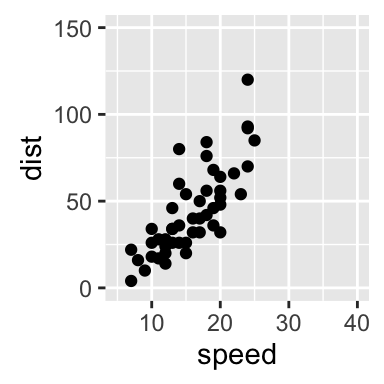
# Nuage de points : limites des axes x et y
sp + xlim(5, 40)+ylim(0, 150)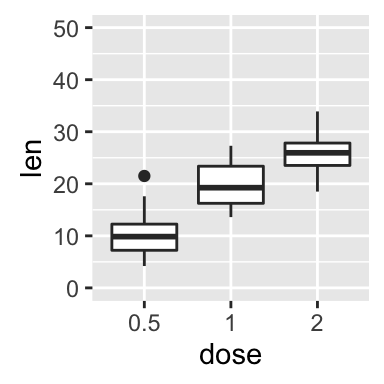
Utiliser la fonction expand_limts()
Notez que, la fonction expand_limits() peut ?tre utilis?e pour :
- d?finir rapidement le point d?intersection des axes x et y ? (0,0)
- changer les limites des axes x et y
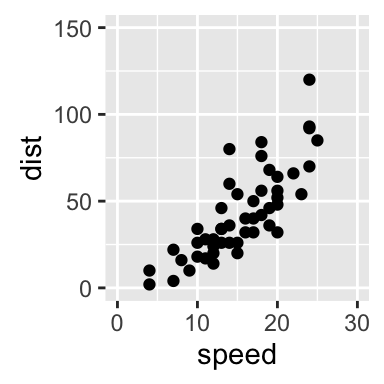
# Fixer l'intersection des axes x et y ? (0,0)
sp + expand_limits(x=0, y=0)
# changer les limites des axes
sp + expand_limits(x=c(0,30), y=c(0, 150))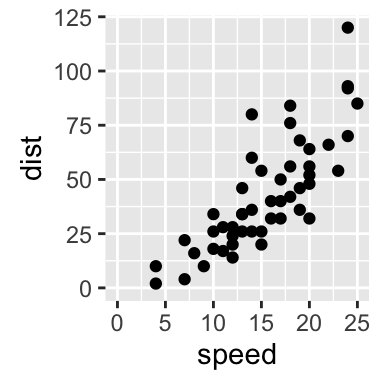
Utiliser les fonctions scale_xx()
Il est aussi possible d?utiliser les fonctions scale_x_continuous() et scale_y_continuous() pour changer les limites des axes x et y, respectivement.
Les formats simplifi?s de ces fonctions sont:
scale_x_continuous(name, breaks, labels, limits, trans)
scale_y_continuous(name, breaks, labels, limits, trans)- name : ?tiquettes (titres) des axes x et y
- breaks : pour contr?ler les graduations et les traits de la grille. Parmi les valeurs possibles, il y a:
- NULL : cache toutes les graduations
- waiver() : graduations par d?faut
- un vecteur de type character ou numeric sp?cifiant les graduations ? afficher
- labels : ?tiquettes des graduations. Les valeurs possibles sont :
- NULL: pas d??tiquettes
- waiver(): ?tiquettes par d?faut
- un vecteur de caract?re ? utiliser comme ?tiquettes des graduations
- limits : un vecteur num?rique sp?cifiant les limites des axes x et y (min, max)
- trans pour la transformation des axes. Les valeurs possibles sont ?log2?, ?log10?, ?
Les fonctions scale_x_continuous() et scale_y_continuous() peuvent ?tre utilis?es comme suit :
# Changer les ?tiquettes et les limites des axes x et y
sp + scale_x_continuous(name="Speed of cars", limits=c(0, 30)) +
scale_y_continuous(name="Stopping distance", limits=c(0, 150))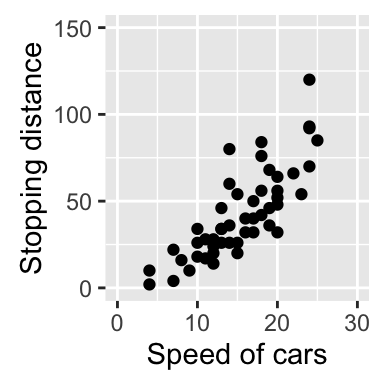
Transformation des axes
Les transformations log et sqrt
Les fonctions pour la transformations des axes sont:
- scale_x_log10(), scale_y_log10() : pour la transformation log10
- scale_x_sqrt(), scale_y_sqrt() : pour la transformation sqrt (racine carr?e)
- scale_x_reverse(), scale_y_reverse() : pour inverser les coordonn?es
- coord_trans(x =?log10?, y=?log10?) : les valeurs possibles pour x et y sont ?log12?, ?log10?, ?sqrt?, ?
- scale_x_continuous(trans=?log2?), scale_y_continuous(trans=?log2?) : une autre valeur possible pour l?argument trans est ?log10?
Ces fonctions peuvent ?tre utilis?es comme suit :
# Nuage de points par d?faut
sp <- ggplot(cars, aes(x = speed, y = dist)) + geom_point()
sp
# Transformation log en utilisant scale_xx()
# valeurs possibles pour trans : 'log2', 'log10','sqrt'
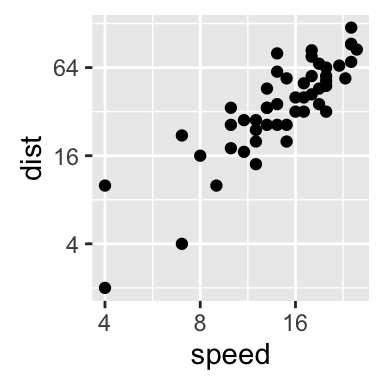
sp + scale_x_continuous(trans='log2') +
scale_y_continuous(trans='log2')
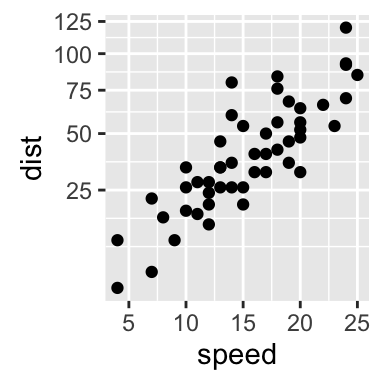
# Transformation sqrt
sp + scale_y_sqrt()
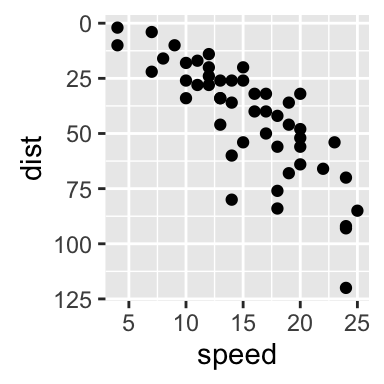
# Inverser les coordonn?es
sp + scale_y_reverse() 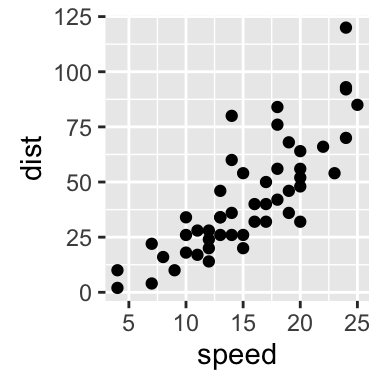
La fonction coord_trans() peut ?tre utilis?e aussi pour la transformation des axes
# Valeurs possibles pour x et y : "log2", "log10", "sqrt", ...
sp + coord_trans(x="log2", y="log2")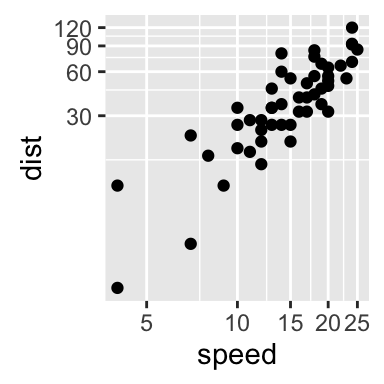
Formater les ?tiquettes des graduations des axes
Les ?tiquettes des graduations peuvent ?tre format?es de mani?re ? montrer des exposants. Le package scales est requis.
# Echelle log2 de l'axe des y (avec espacement visuelle ?gale)
library(scales)
sp + scale_y_continuous(trans = log2_trans())
# Montrer des exposants
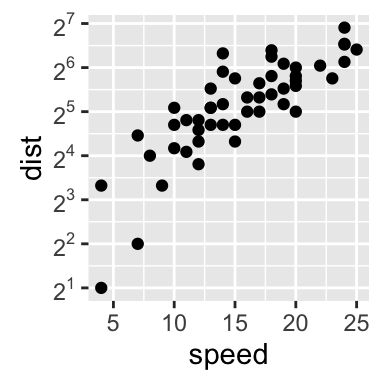
sp + scale_y_continuous(trans = log2_trans(),
breaks = trans_breaks("log2", function(x) 2^x),
labels = trans_format("log2", math_format(2^.x)))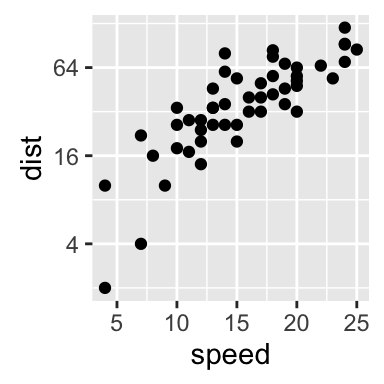
Notez que beaucoup de fonctions de transformation sont disponibles en utilisant le package scales: log10_trans(), sqrt_trans(), etc. Utiliser la commande help(trans_new) pour une liste compl?te.
Formater les ?tiquettes des axes :
library(scales)
# Pourcentage
sp + scale_y_continuous(labels = percent)
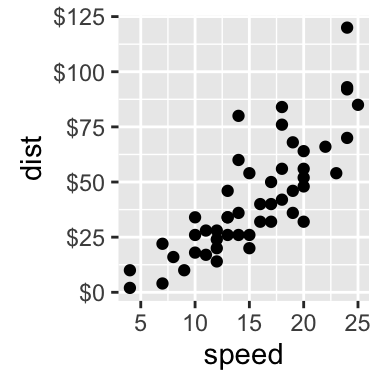
# dollar
sp + scale_y_continuous(labels = dollar)
# scientifique
sp + scale_y_continuous(labels = scientific)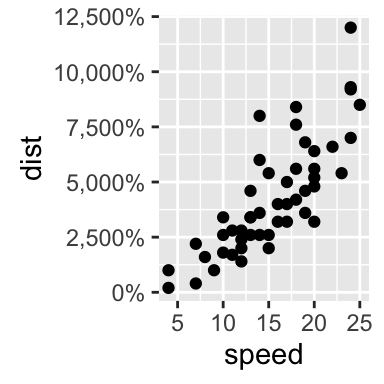
Afficher des graduations log
Il est possible d?ajouter des graduations log en utilisant la fonction annotation_logticks().
Notez que ce type de graduations ? un sens uniquement pour log base 10.
Le jeu de donn?es Animals, dans le package MASS, est utilis? :
library(MASS)
head(Animals)## body brain
## Mountain beaver 1.35 8.1
## Cow 465.00 423.0
## Grey wolf 36.33 119.5
## Goat 27.66 115.0
## Guinea pig 1.04 5.5
## Dipliodocus 11700.00 50.0La fonction annotation_logticks() peut ?tre utilis?e comme suit :
library(MASS) # Acc?der aux donn?es Animals
library(scales) # Acc?der aux fonctions de transformations
# les axes x et y sont transform?s et format?s
p2 <- ggplot(Animals, aes(x = body, y = brain)) + geom_point() +
scale_x_log10(breaks = trans_breaks("log10", function(x) 10^x),
labels = trans_format("log10", math_format(10^.x))) +
scale_y_log10(breaks = trans_breaks("log10", function(x) 10^x),
labels = trans_format("log10", math_format(10^.x))) +
theme_bw()
# graphique log-log sans les ?tiquettes de graduations
p2
# Montrer les ?tiquettes de graduations
p2 + annotation_logticks() 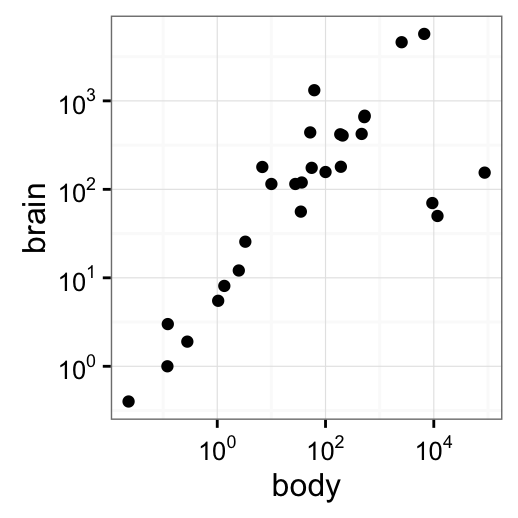
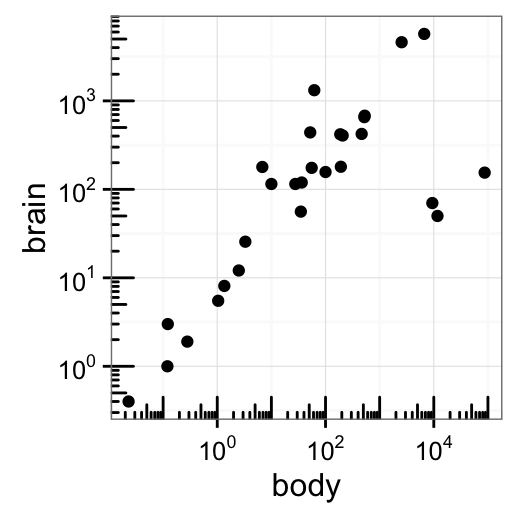
Notez que, par d?faut les graduations log sont en bas et ? gauche.
Pour sp?cifier le c?t? des graduations :
# Graduations ? gauche et ? droite
p2 + annotation_logticks(sides="lr")
# Tous les c?t?s
p2+annotation_logticks(sides="trbl")Les valeurs possibles pour l?argument sides sont :
- t : en haut
- r : ? droite
- b : en bas
- l : ? gauche
- la combinaison de t, r, b et l
Formater les axes pour des dates
Les fonctions scale_x_date() et scale_y_date() sont utilis?es.
Exemple de donn?es
Cr?er des donn?es de s?rie temporelle
df <- data.frame(
date = seq(Sys.Date(), len=100, by="1 day")[sample(100, 50)],
price = runif(50)
)
df <- df[order(df$date), ]
head(df)## date price
## 22 2016-09-21 0.12484956
## 10 2016-09-22 0.39837832
## 40 2016-09-23 0.52704801
## 45 2016-09-25 0.05024957
## 32 2016-09-27 0.98075239
## 49 2016-09-28 0.64052733Graphe avec des dates
dp <- ggplot(data=df, aes(x=date, y=price)) + geom_line()
dp
Formater les ?tiquettes de graduations
Charger le package scales pour le formatage.
library(scales)
# Format : month/day
dp + scale_x_date(labels = date_format("%m/%d")) +
theme(axis.text.x = element_text(angle=45))
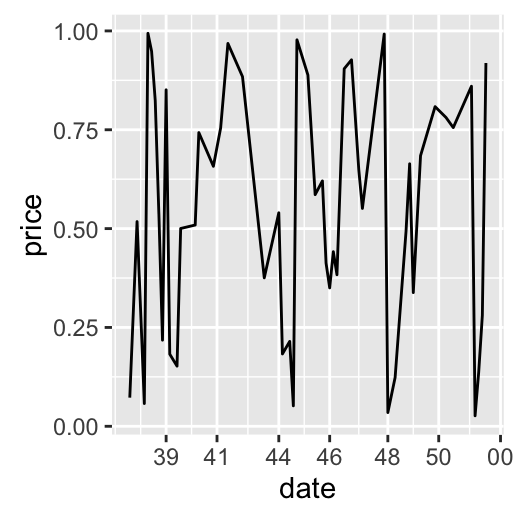
# Format : Week
dp + scale_x_date(labels = date_format("%W"))
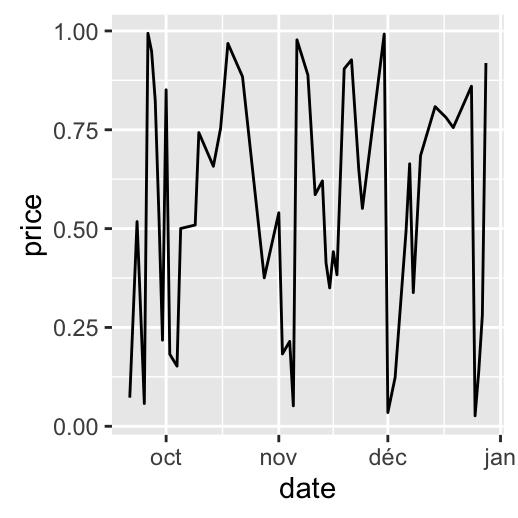
# Months only
dp + scale_x_date(breaks = date_breaks("months"),
labels = date_format("%b"))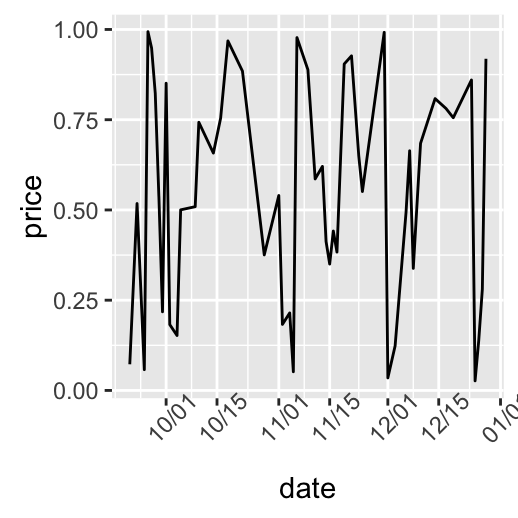
Notez que, depuis ggplot2 v2.0.0, les ?chelles date et datetime poss?dent les param?tres date_breaks, date_minor_breaks et date_labels. Vous n?avez donc plus besoin de scales::date_breaks() ou de scales::date_format().
Limite des axes pour des dates
Le jeu de donn?es economic (package ggplot2) est utilis? :
head(economics)## date pce pop psavert uempmed unemploy
## 1 1967-07-01 507.4 198712 12.5 4.5 2944
## 2 1967-08-01 510.5 198911 12.5 4.7 2945
## 3 1967-09-01 516.3 199113 11.7 4.6 2958
## 4 1967-10-01 512.9 199311 12.5 4.9 3143
## 5 1967-11-01 518.1 199498 12.5 4.7 3066
## 6 1967-12-01 525.8 199657 12.1 4.8 3018Cr?er le graphique de psavert en fonction de date:
- date : Mois de collection des donn?es
- psavert : ?personal savings rate?
# Graphe avec des dates
dp <- ggplot(data=economics, aes(x=date, y=psavert)) + geom_line()
dp
# Limites des axes c(min, max)
min <- as.Date("2002-1-1")
max <- max(economics$date)
dp+ scale_x_date(limits = c(min, max))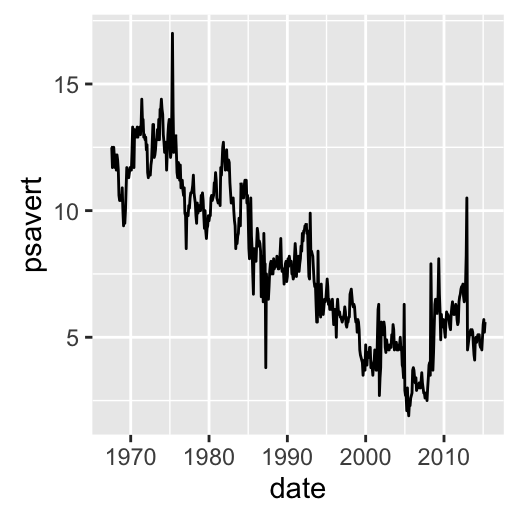
Aller plus loin
Voir aussi les fonctions scale_x_datetime() et scale_y_datetime() pour dessiner des graphiques contenant des dates et le temps.
Infos
Cette analyse a ?t? r?alis?e avec le logiciel R (ver. 3.2.4) et le package ggplot2 (ver. 2.1.0)
Show me some love with the like buttons below... Thank you and please don't forget to share and comment below!!
Montrez-moi un peu d'amour avec les like ci-dessous ... Merci et n'oubliez pas, s'il vous plaît, de partager et de commenter ci-dessous!
Recommended for You!
Click to follow us on Facebook:
Comment this article by clicking on "Discussion" button (top-right position of this page)







